Epigenética en Diabetes Mellitus
Muchas modificaciones epigenéticas parecen estar influidas por factores ambientales. En este sentido, la plasticidad del genoma permitiría una mejor adaptación al medio. Los principales mecanismos epigenéticos que regulan la expresión de los genes son:
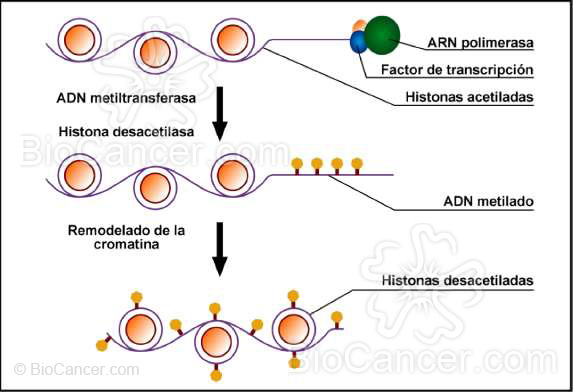
• La metilación del ADN en citosinas seguidas por guaninas (dinucleótidosCpGs). Estos dinucleótidos son abundantes en las regiones promotoras de muchos genes, y su hipermetilación suele acompañarse de una disminución de la expresión génica.
• Diversas modificaciones covalentes en aminoácidos terminales de las histonas, incluyendo, entre otras, acetilación y metilación, que son moduladas por enzimas como las acetiltransferasas (HATs), metiltransferasas (HMTs), y desacetilasas (HDACs). Estas modificaciones postranscripcionales parecen afectar a la expresión génica a través de alteraciones en el grado de compactación o dificultando el acceso de los factores de transcripción al ADN.
• Los RNAs no codificantes, entre los que destacan los micro RNAs (miRNAs), que regulan post-transcripcionalmente la expresión de genes mediante su emparejamiento con la región no traducida 3 del ARN mensajero y la consiguiente degradación de los transcritos.